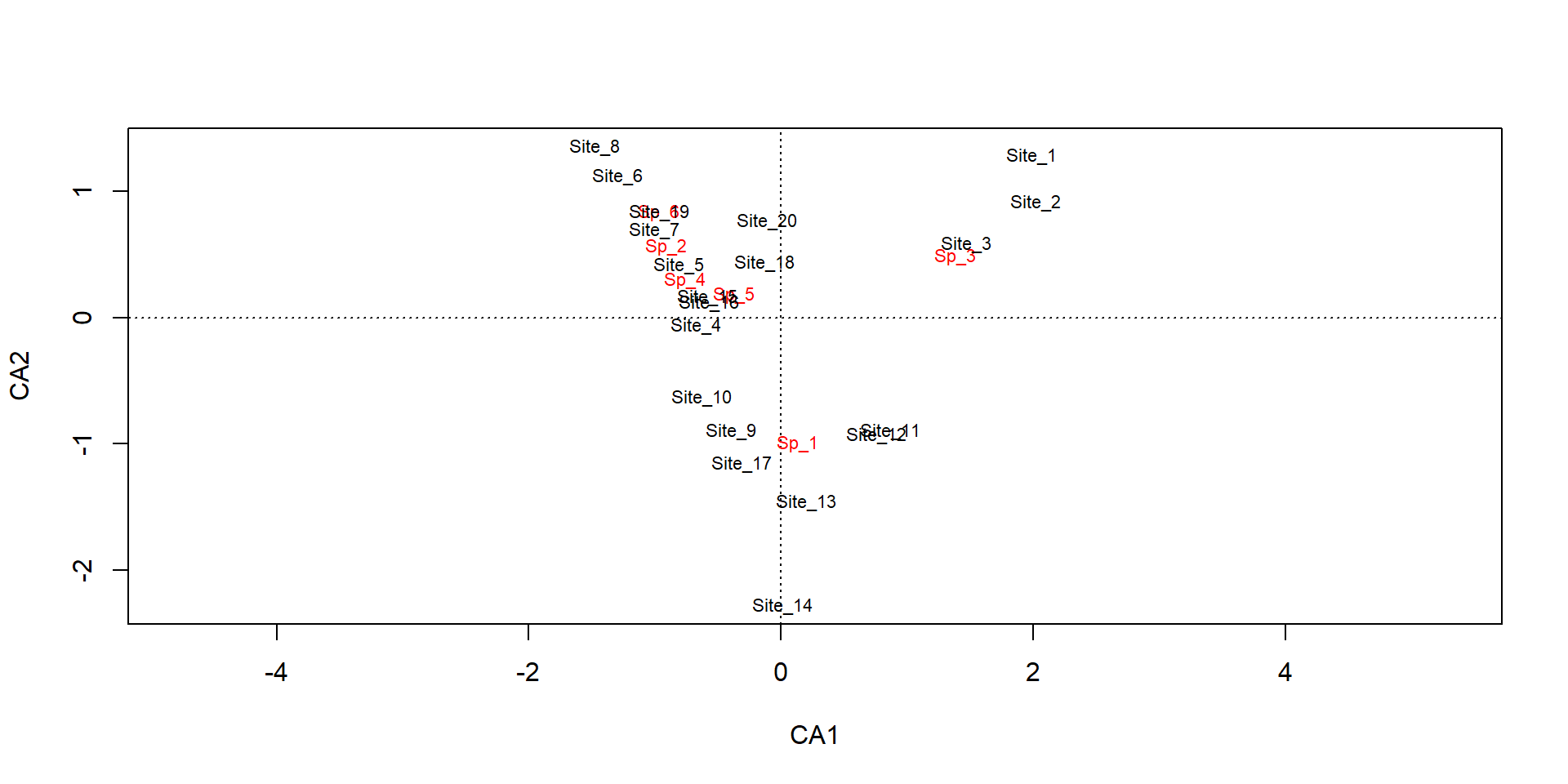
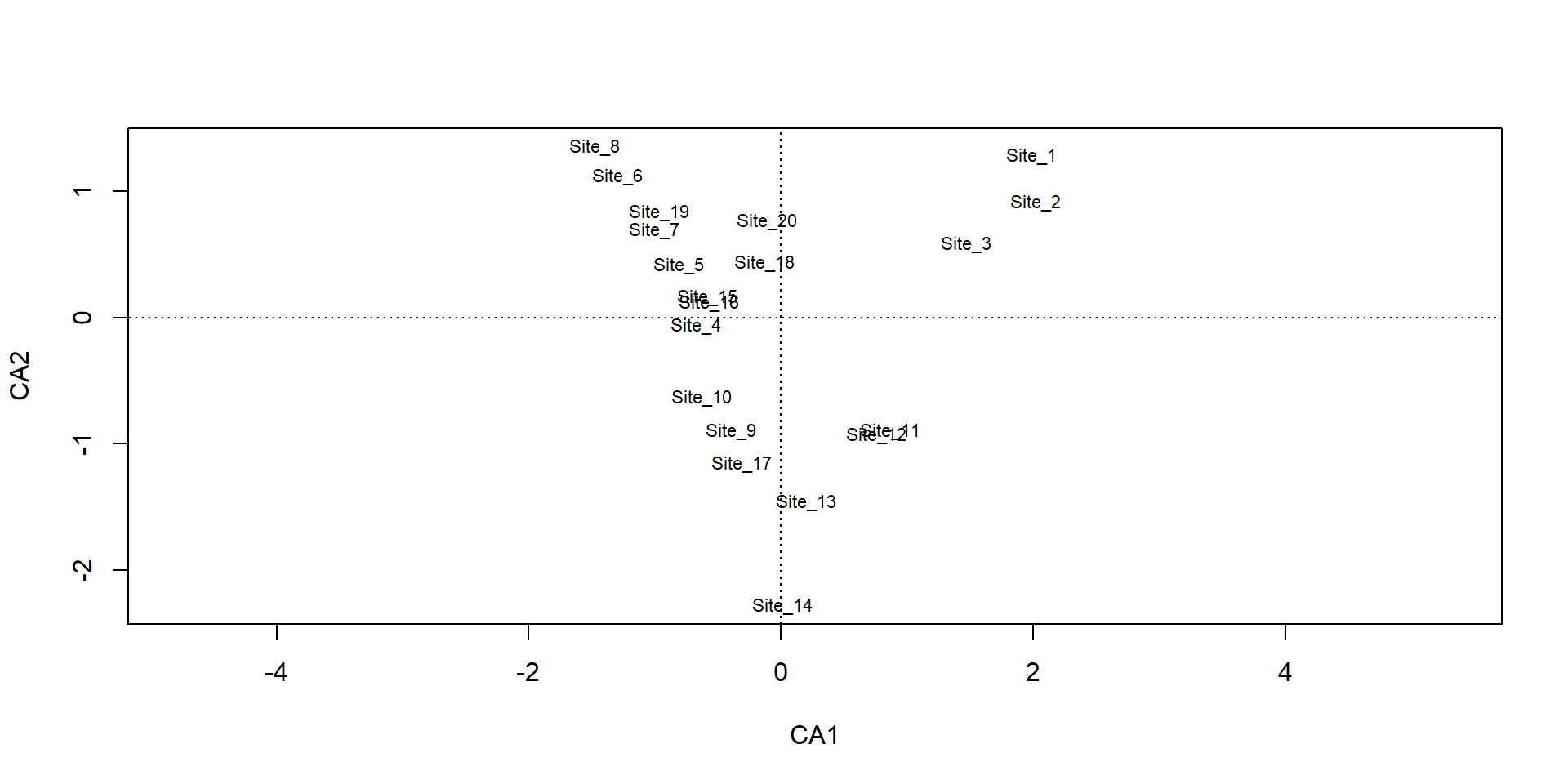
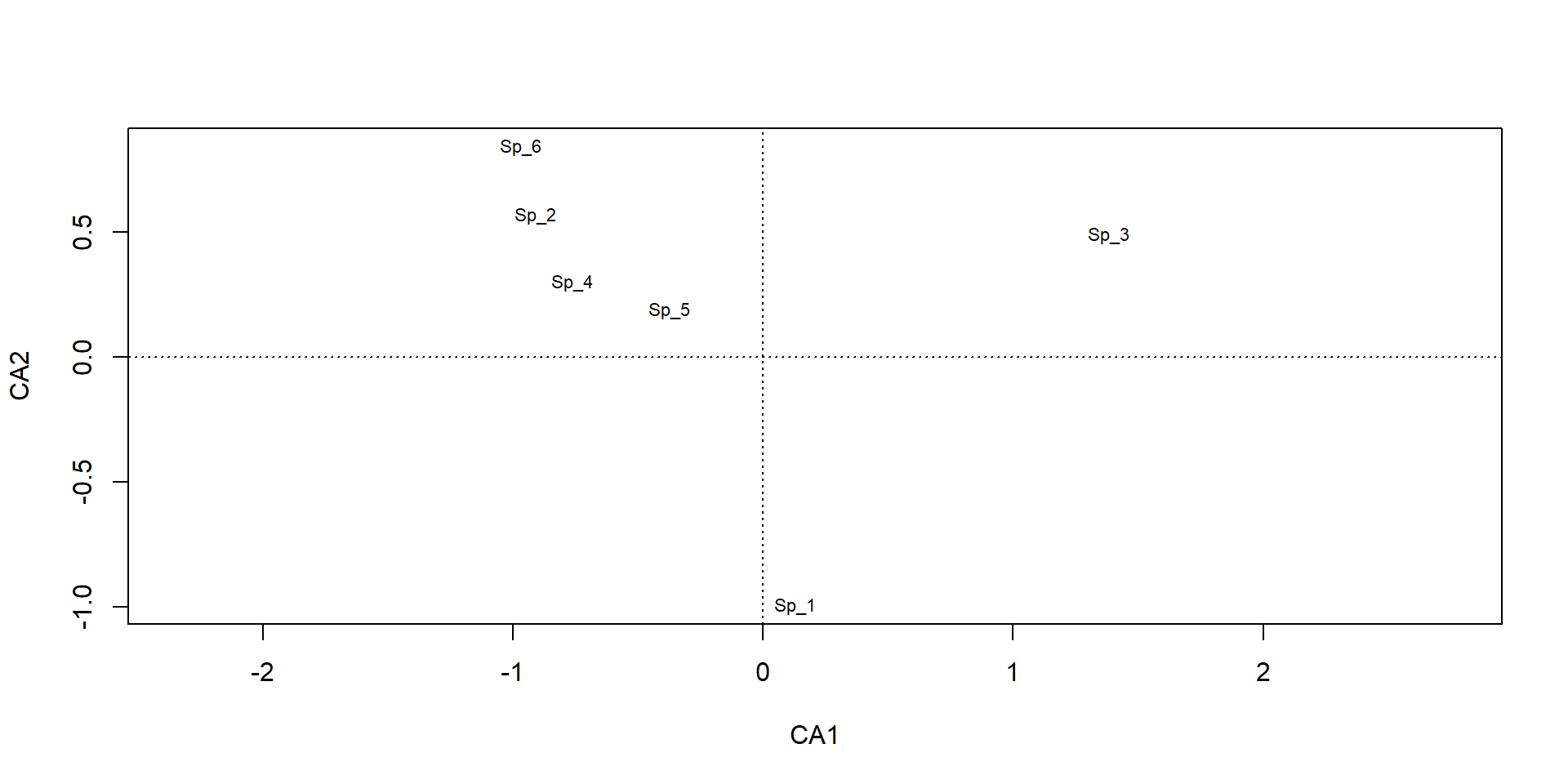
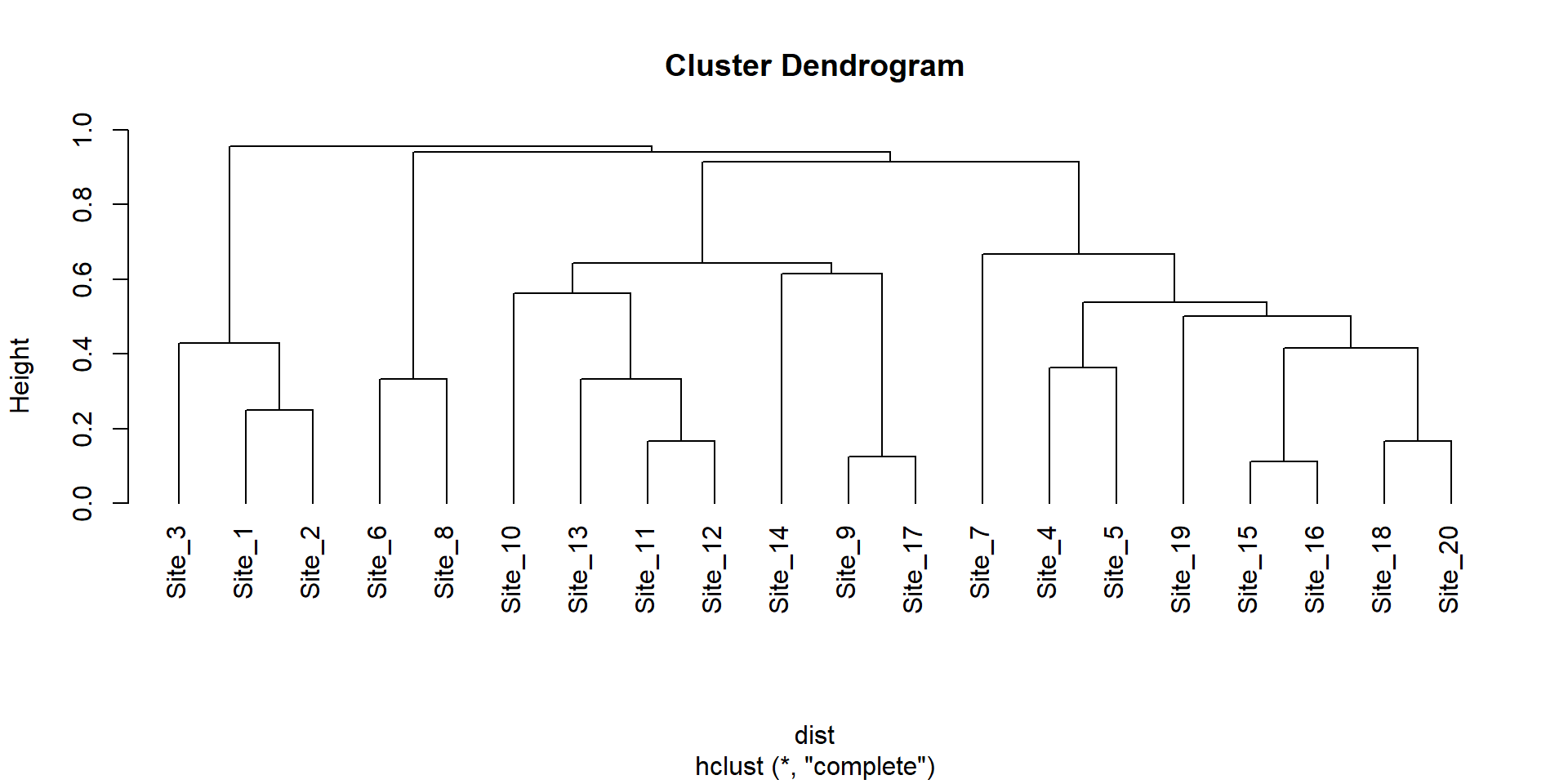
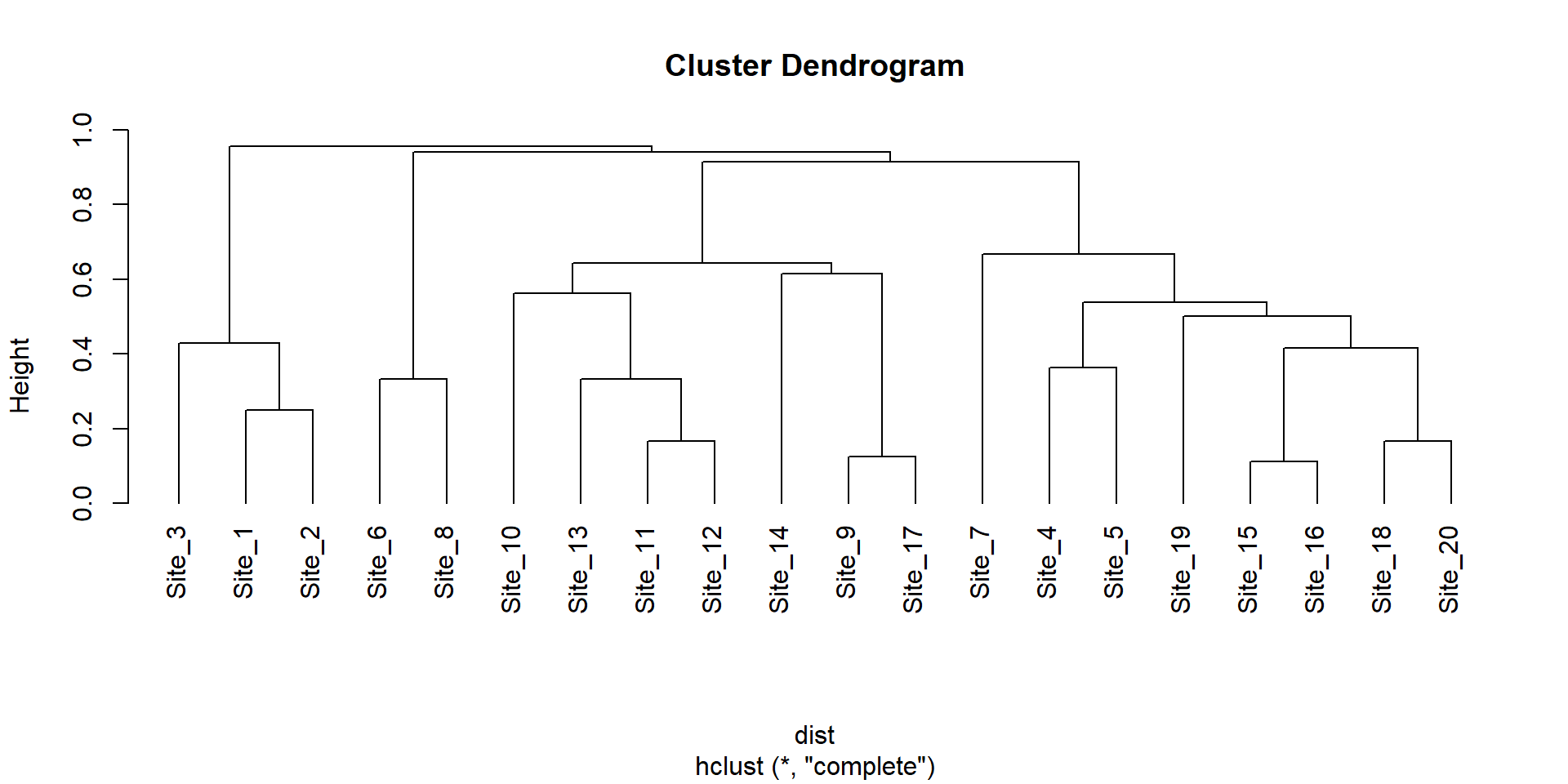
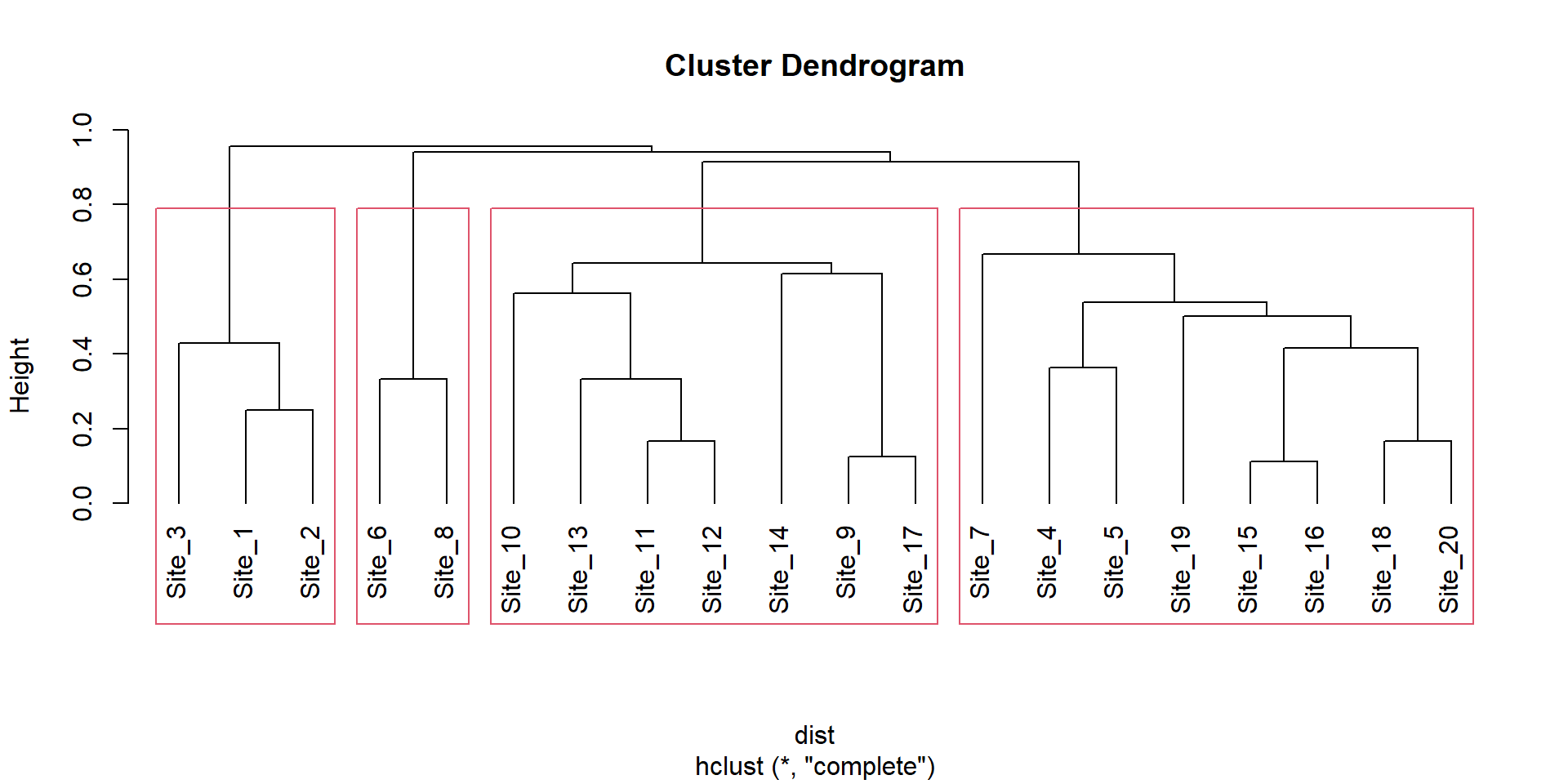
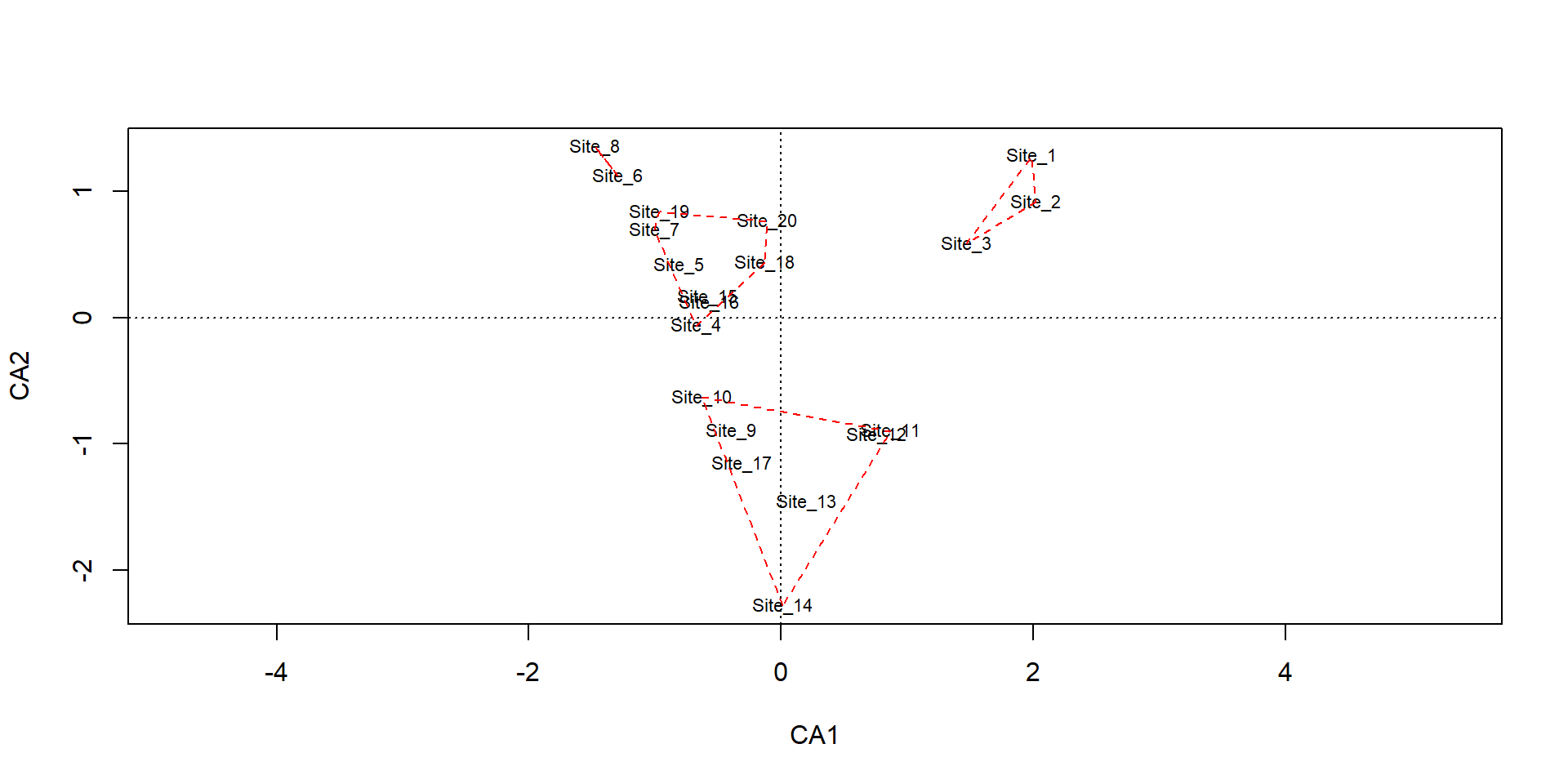
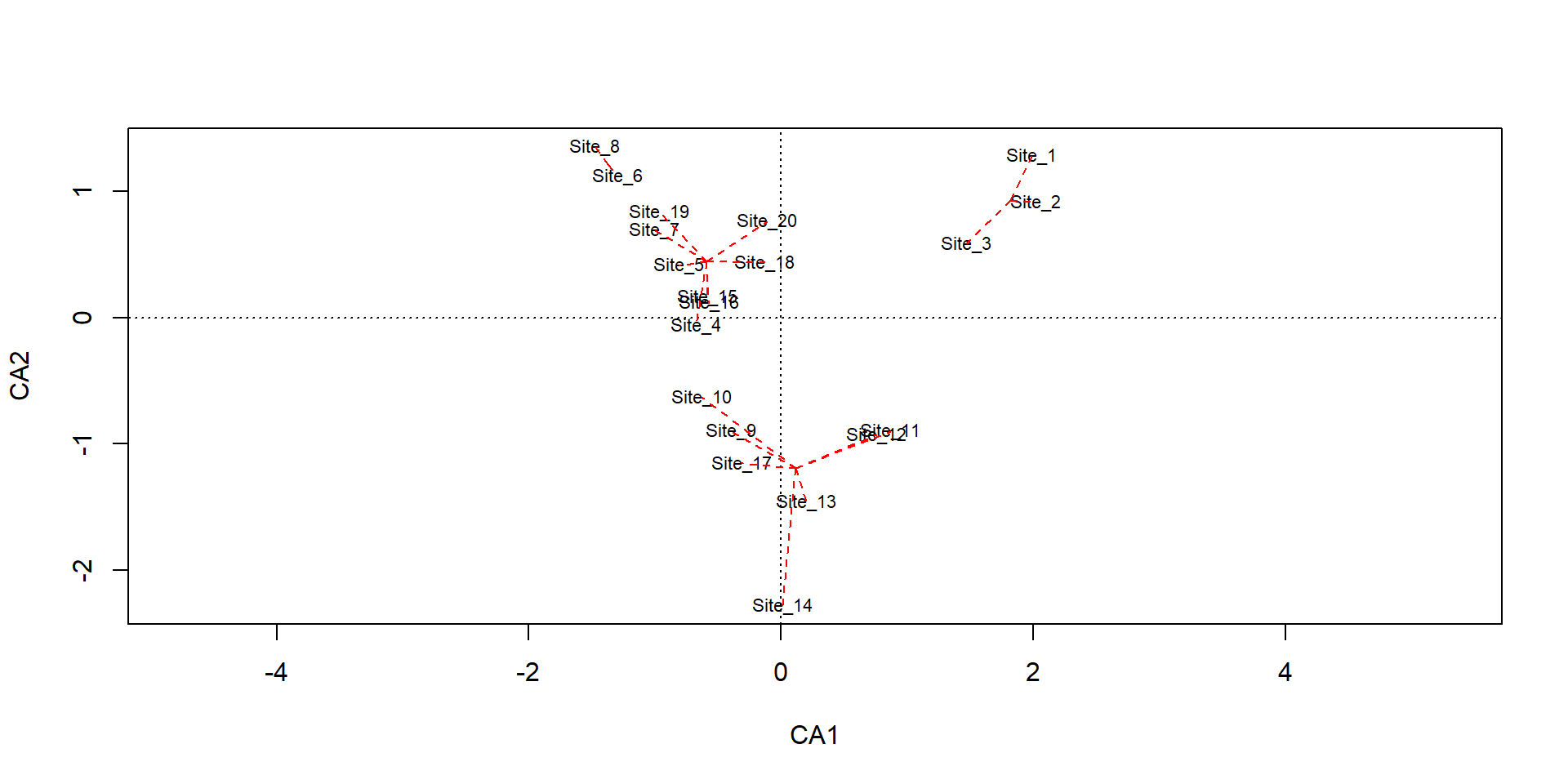
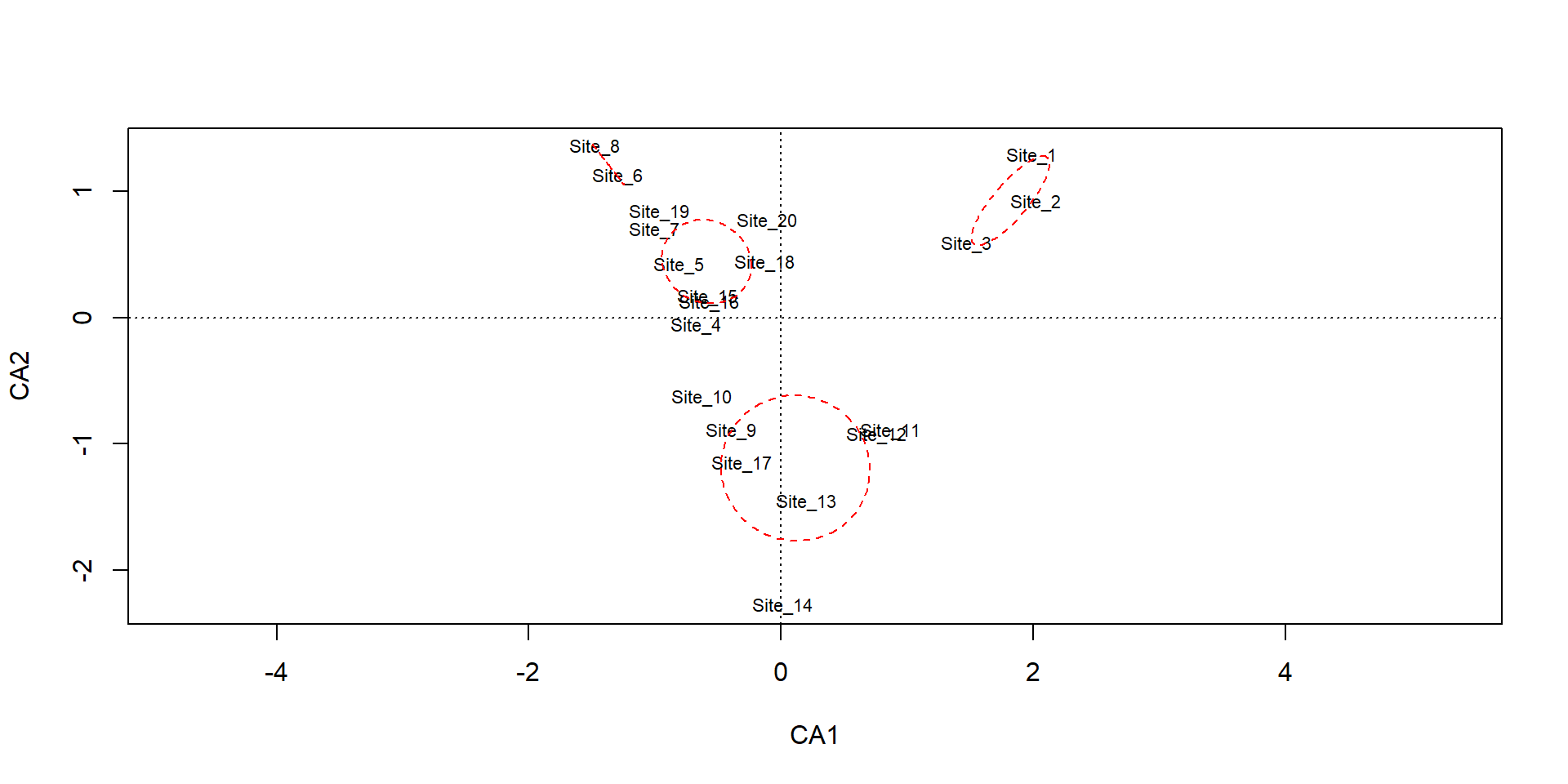
Sp_1 Sp_2 Sp_3 Sp_4 Sp_5 Sp_6
Site_1 0 0 9 0 2 0
Site_2 1 0 8 0 1 0
Site_3 2 1 7 0 1 0
Site_4 2 1 0 1 5 0
Site_5 1 2 0 0 6 0
Site_6 0 4 0 2 3 0 v1 v2 v3
Site_1 5.1789918 -0.4380167 2.8471131
Site_2 4.4714402 1.8806640 -0.3560559
Site_3 2.9379526 -0.3054643 3.2177157
Site_4 0.7354044 -1.9925002 1.2593483
Site_5 2.6035489 -2.5324108 1.9391404
Site_6 0.6951173 2.8438965 -1.2821121