# Ejercicio 1.
# Parte 1
seq(1, 40, by = 2)
# Parte 2
seq(1, 40, by = -2)
# Este código da error porque especifiqué algo mal :)Análisis espaciales y multivariantes con R (AEMeR) aplicados a estudios de biodiversidad
Ejercicios de manipulación de datos espaciales
Los ejercicios tendréis que entregarlos para la evaluación del curso. Para ello, crea un script de R con RStudio. Ponle el nombre ejercicios_t2_[nombre]_[apellido].R. Dicho script deberá estar organizado con comentarios en castellano (poniendo una almohadilla # delante) y justo detrás el código necesario para realizar el ejercicio (ver ejemplo debajo). Además, si el ejercicio pide reflexionar sobre el resultado use los comentarios (con la #) para indicar sus reflexiones debajo del código. Una vez los tengáis completos podéis mandármelos por email.
Parte 1. Trabajando con datos vectoriales
Funciones útiles
vect()
Se usa para generar o leer datos vectoriales. Es muy flexible en su forma de funcionar, por lo que tendrás que leer bien la documentación para ver que argumentos necesita.
gadm()
Esta función del paquete geodata se usa para cargar datos de los límites administrativos de los distintos países del planeta.
project()
Proyecta las coordenadas de los objetos espaciales a un sistema de coordenadas diferentes. Para conseguir los códigos EPSG de distintos sistemas de proyección podéis usar la siguiente página: https://epsg.io/
[]
Los corchetes sirven para extraer elementos de ciertas posiciones en los objetos de R. Se pueden usar también con los objetos espaciales
==
Se usa para realizar comparaciones y generar un vector de valores lógicos que indican cuando se cumple una condición de igualdad.
writeVector
Esta función se usa para guardar objetos vectoriales en el disco duro del ordenador.
Ejercicios
1. Cargue los límites administrativos de los departamentos de Argentina en un objeto llamado departamentos y filtre los departamentos para retener sólo los departamentos de la provincia de "Buenos Aires".
library(geodata)
departamentos <- gadm("ARG", 2, path = ".")
i <- which(departamentos$NAME_1 == "Buenos Aires")
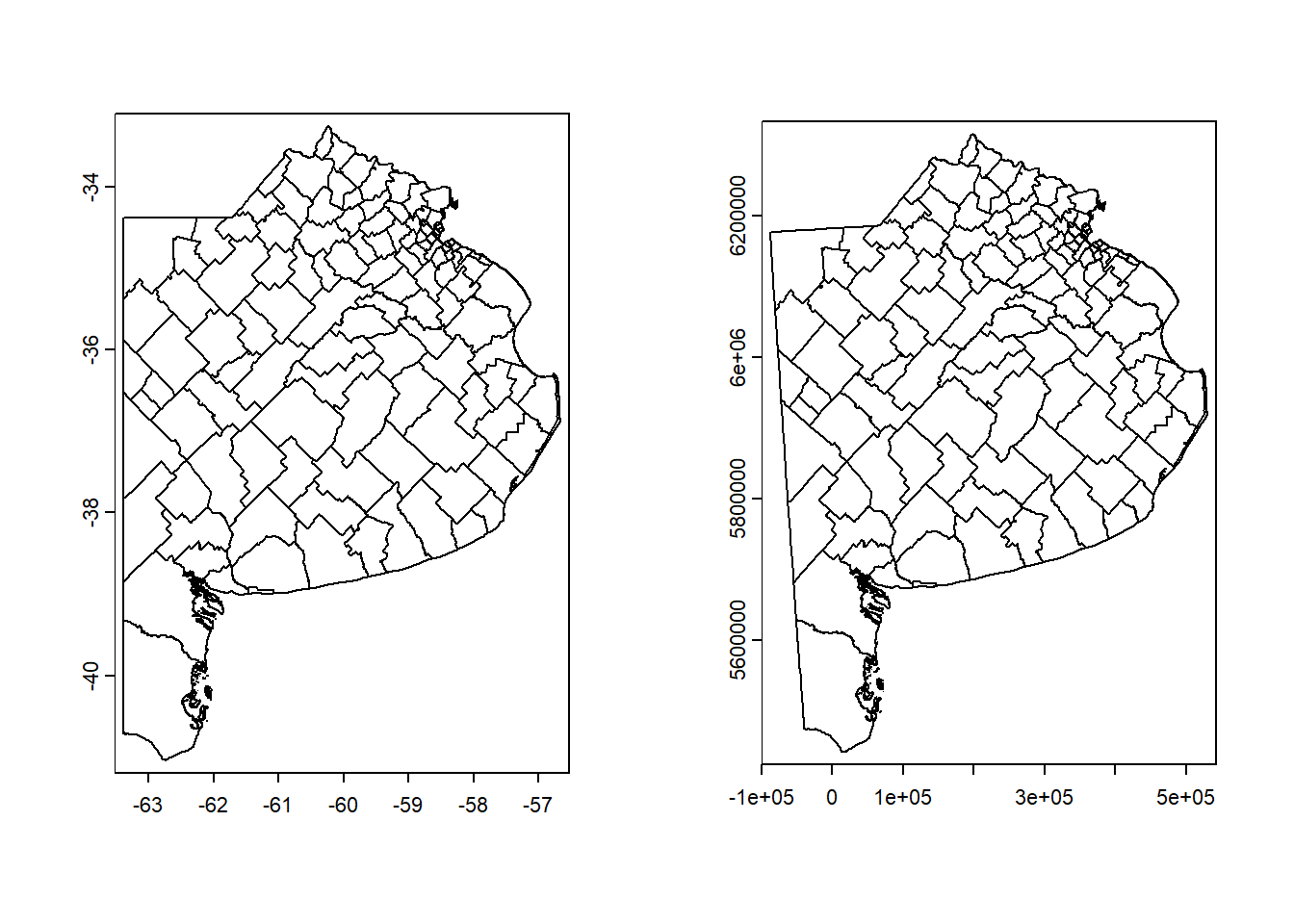
ba <- departamentos[i]2. Ahora, proyecte los datos de estos municipios desde el sistema de coordenadas en longitud-latitud y WGS84 al sistema UTM21 sur. Para ello necesitaréis buscar el código EPSG en https://epsg.io.
ba_utm <- project(ba, "epsg:32721")3. Guarda el objeto vectorial de Buenos Aires con las coordenadas en UTM21s como un fichero geopackage nuevo, llamado BuenosAires.gpkg
writeVector(ba_utm, "BuenosAires.gpkg", overwrite = TRUE)4. Finalmente, dibuje dos mapas uno con el mapa de la provincia de Buenos Aires en longitud-latitud (WGS84) y el otro en UTM21s. Por último .
Parte 2. Trabajando con datos ráster
Funciones útiles
rast()
Se utiliza para generar o cargar datos ráster. Es muy flexible también en su forma de usarse, por lo que tendrás que tener cuidad con los argumentos que usas con ella.
worldclim_country()
Esta función del paquete geodata se usa para cargar datos climáticos de cualquier país del planeta. Los datos proceden del proyecto WorldClim.
project()
Se usa para proyectar tanto datos vectoriales como raster.
writeRaster()
Se usa para guardar objetos ráster en el disco duro del ordenador.
Ejercicios
1. Carga las variables bioclimáticas (v.gr. var = "bio") del proyecto WorldClim a 0.5 minutos de grado de resolución.
library(geodata)
arg_bio <- worldclim_country("ARG", var = "bio", path = ".")2. Fíjese el nombre tan largo y poco intuitivo de los nombres de las distintas capas (puede consultarlo usando el siguiente código names(bio)). Cambie el nombre de las variables por algo más corto e intuitivo (v.gr. “bio1”, “bio2”, etc.).
names(arg_bio)
names(arg_bio) <- paste0("bio", 1:19)3. Revisa el CRS de los datos que has descargado. ¿Coinciden con los datos que has generado como resultado de los ejercicios de la Parte 1? Si no es así, proyecta los datos raster para que coincidan con esos datos y mantengan una resolución exacta de 1000 metros.
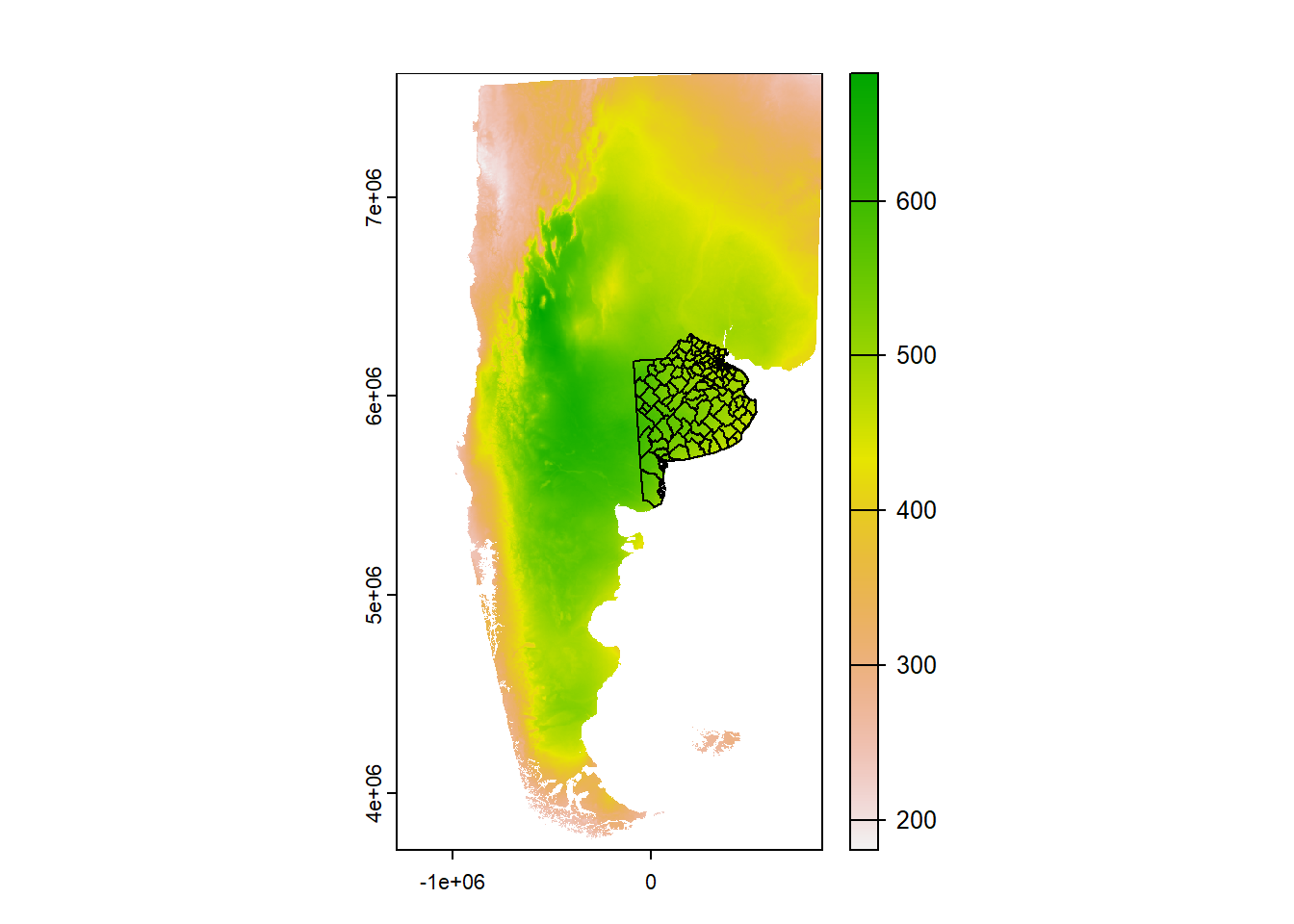
arg_bio_utm <- project(arg_bio, "epsg:32721", res = 1000)4. Guarde los dos objetos ráster (original y resultante) como un fichero en el disco duro de su ordenador, de modo que pueda reutilizarlos más adelante cuando los vuelva a necesitar.
writeRaster(arg_bio, "arg_bio.tif", overwrite = TRUE)
writeRaster(arg_bio_utm, "arg_bio_utm.tif", overwrite = TRUE) 5. Finalmente, para cerciorarnos que todo esté correcto, dibuje el mapa de la variable "bio4" en UTM19s con los polígonos de los municipios de la provincia de “Buenos Aires” superpuestos.
Parte 3. Manipulación datos ráster y vectoriales
Funciones útiles
expanse()
Se usa para calcular la superficie de cada uno de los polígonos en una capa vectorial.
aggregate()
Sirve para agregar formas vectoriales que tienen el mismo valor en alguno de los campos de los atributos, el cual se usa como criterio de agregación. También se usa para agregar los valores de varios píxeles contiguos en datos ráster.
mask()
Se usa para eliminar la información de un ráster que queda fuera de la superficie delimitada por los polígonos de una capa vectorial.
Ejercicios
1. Añade una columna de atributos a la capa de los municipios de “Buenos Aires” en la que se recoja el área (superficie) de cada municipio. Posteriormente, dibuja un mapa de los municipios en los que se pueda observar con colores diferentes el área de cada uno de ellos. ¿Hay un patrón geográfico en el tamaño de los municipios de la provincia?
ba$area <- expanse(ba)
plot(ba, "area", type = "interval")2. Agrega todos los municipios de la provincia de “Buenos Aires” para generar un polígono con la forma de la provincia. Dibuja el mapa del ejercicio anterior y adiciona el borde de la provincia con los colores de la bandera argentina.
ba_borde <- aggregate(ba, "NAME_1")
plot(ba, "area", type = "interval")
plot(ba_borde, lwd = 4, border = "cyan", add = TRUE)
plot(ba_borde, lwd = 2, border = "white", add = TRUE)3. Recorta el objeto ráster con las variables bioclimáticas de Argentina para quedarte sólo con los datos de la provincia de “Buenos Aires”. Dibuja el mapa de la variable bio4 de dichos datos. ¿Ves algo raro?
ba_bio <- mask(arg_bio, ba)
plot(ba_bio[["bio4"]])4. Usa el siguiente código x <- trim(x) (donde x es el nombre del objeto resultante del ejercicio anterior) y vuelve a dibujar el mapa raster resultante. ¿Que diferencia observas con el mapa del ejercicio anterior? ¿Qué crees que hace la función?
ba_bio <- trim(ba_bio)
plot(ba_bio[["bio4"]])